1 引言
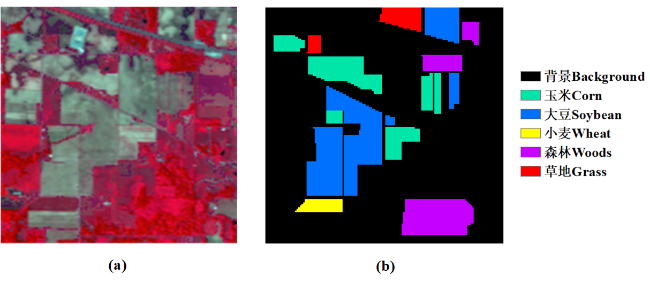
2 数据与方法
2.1 数据及数据集设置
2.2 光谱特征提取方法
2.3 空间特征提取方法
3 结果与分析
3.1 混合光谱特征提取方法的性能
表1 PCA和PCA_ScatterMatrix方法在数据集2上获得最高分类正确率时所选取的主成分序号Table 1 Selected principal components when PCA and PCA_ScatterMatrix got the highest overall accuracy on data set 2 |
| 方法 | 选取的主成分的序号 |
|---|---|
| PCA | 1,2,3,4,5,6 |
| PCA_ScatterMatrix | 1,2,3,4,5,6,7,8,10,11,12,13,68 |
表2 在数据集2上各光谱特征提取方法的总体分类正确率、生产者精度和Kappa系数的比较Table 2 Performance of the feature extraction methods investigated overall classification accuracy, producer's accuracy and Kappa coefficient on data set 2 |
| 分类方法 | 特征数目 | 总体分类正确率 | Kappa系数 | 生产者精度 | ||||
|---|---|---|---|---|---|---|---|---|
| 玉米 | 大豆 | 小麦 | 森林 | 草地 | ||||
| PCA | 6 | 0.840 | 0.764 | 0.807 | 0.824 | 0.982 | 0.932 | 0.712 |
| ICA | 15 | 0.827 | 0.746 | 0.822 | 0.822 | 1.000 | 0.843 | 0.792 |
| PCA_ScatterMatrix | 13 | 0.865 | 0.799 | 0.820 | 0.878 | 0.982 | 0.909 | 0.746 |
| LDA | 4 | 0.824 | 0.743 | 0.815 | 0.784 | 0.982 | 0.905 | 0.856 |
3.2 光谱特征提取方法与空间特征提取方法的性能比较
表3 在数据集1上光谱特征和空间特征在高光谱影像植被分类中的性能Table 3 Performance of the spectral and spatial features of hyperspectral image vegetation classification on data set 1 |
| 方法 | 波段数目 | 总体正确率 | Kappa系数 | 生产者精度 | ||||
|---|---|---|---|---|---|---|---|---|
| 玉米 | 大豆 | 玉米 | 森林 | 草地 | ||||
| JMI | 33 | 0.778 | 0.847 | 0.846 | 0.830 | 0.990 | 0.904 | 0.703 |
| mRMR | 40 | 0.725 | 0.603 | 0.632 | 0.742 | 0.905 | 0.813 | 0.591 |
| CMIM | 32 | 0.837 | 0.764 | 0.791 | 0.836 | 0.990 | 0.900 | 0.752 |
| DISR | 40 | 0.858 | 0.795 | 0.860 | 0.849 | 0.981 | 0.882 | 0.780 |
| JM | 19 | 0.803 | 0.718 | 0.808 | 0.758 | 0.971 | 0.882 | 0.773 |
| GLCM | 8 | 0.857 | 0.798 | 0.864 | 0.803 | 1.000 | 0.915 | 0.990 |
| Gabor | 80 | 0.955 | 0.935 | 0.980 | 0.918 | 1.000 | 0.986 | 1.000 |
| Morph | 20 | 0.904 | 0.860 | 0.870 | 0.884 | 0.990 | 0.967 | 0.941 |
表4 在数据集2上光谱特征和空间特征在高光谱影像植被分类中的性能Table 4 Performance of the spectral and spatial features of hyperspectral image vegetation classification on data set 2 |
| 方法 | 波段数目 | 总体正确率 | Kappa系数 | 生产者精度 | ||||
|---|---|---|---|---|---|---|---|---|
| 玉米 | 大豆 | 小麦 | 森林 | 草地 | ||||
| JMI | 40 | 0.863 | 0.798 | 0.823 | 0.861 | 0.982 | 0.913 | 0.839 |
| mRMR | 40 | 0.757 | 0.644 | 0.708 | 0.732 | 0.945 | 0.883 | 0.623 |
| CMIM | 32 | 0.846 | 0.773 | 0.784 | 0.844 | 0.982 | 0.917 | 0.839 |
| DISR | 23 | 0.844 | 0.768 | 0.760 | 0.852 | 0.982 | 0.938 | 0.750 |
| JM | 24 | 0.856 | 0.788 | 0.843 | 0.843 | 1.000 | 0.899 | 0.818 |
| GLCM | 8 | 0.874 | 0.818 | 0.894 | 0.825 | 1.000 | 0.921 | 0.987 |
| Gabor | 80 | 0.967 | 0.951 | 0.984 | 0.944 | 1.000 | 0.987 | 1.000 |
| Morph | 20 | 0.926 | 0.890 | 0.871 | 0.928 | 1.000 | 0.970 | 0.987 |