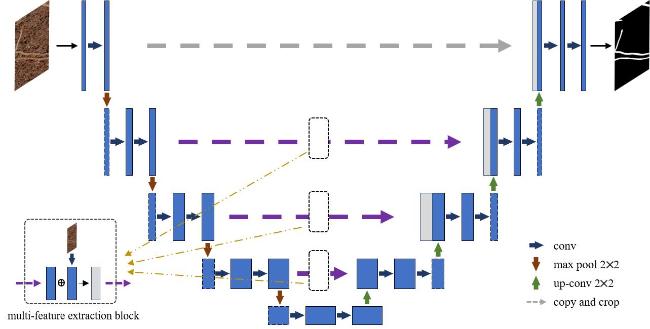
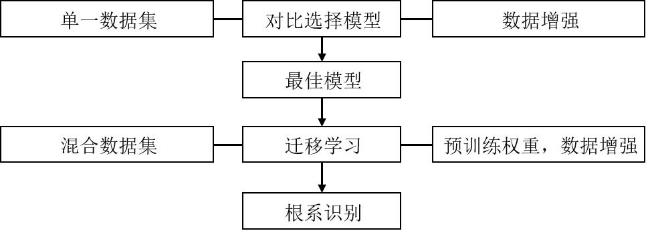
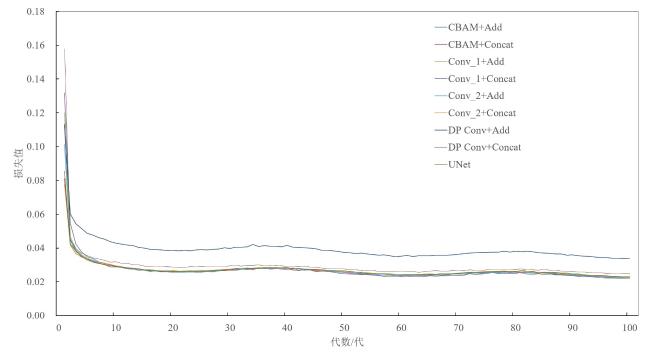
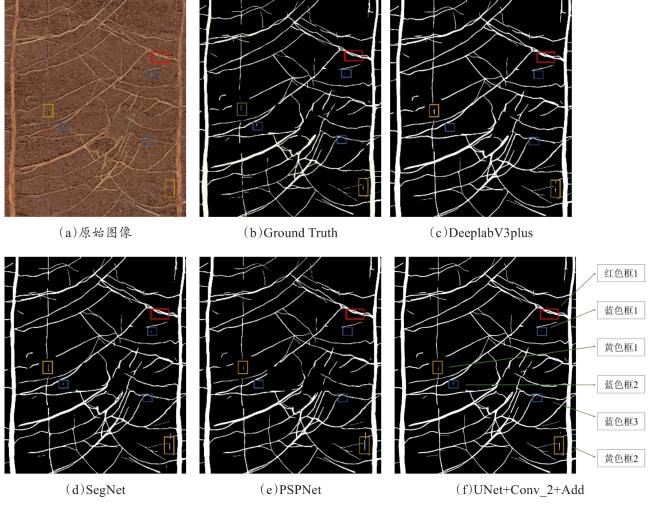
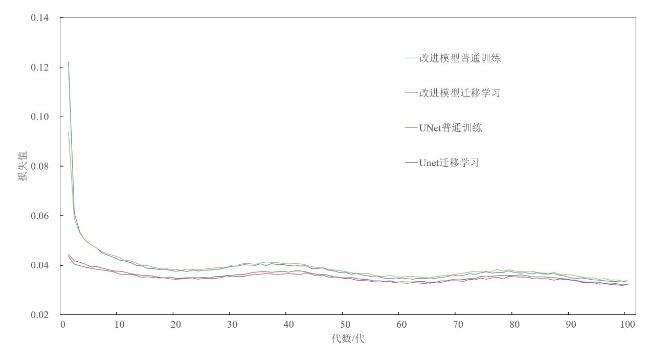
[Objective] The root system is an important component of plant composition, and its growth and development are crucial for plants. Root image segmentation is an important method for obtaining root phenotype information and analyzing root growth patterns. Research on root image segmentation still faces difficulties, because of the noise and image quality limitations, the intricate and diverse soil environment, and the ineffectiveness of conventional techniques. This paper proposed a multi-scale feature extraction root segmentation algorithm that combined data augmentation and transfer learning to enhance the generalization and universality of the root image segmentation models in order to increase the speed, accuracy, and resilience of root image segmentation. [Methods] Firstly, the experimental datasets were divided into a single dataset and a mixed dataset. The single dataset acquisition was obtained from the experimental station of Hebei Agricultural University in Baoding city. Additionally, a self-made RhizoPot device was used to collect images with a resolution pixels of 10,200×14,039, resulting in a total of 600 images. In this experiment, 100 sheets were randomly selected to be manually labeled using Adobe Photoshop CC2020 and segmented into resolution pixels of 768×768, and divided into training, validation, and test sets according to 7:2:1. To increase the number of experimental samples, an open source multi-crop mixed dataset was obtained in the network as a supplement, and it was reclassified into training, validation, and testing sets. The model was trained using the data augmentation strategy, which involved performing data augmentation operations at a set probability of 0.3 during the image reading phase, and each method did not affect the other. When the probability was less than 0.3, changes would be made to the image. Specific data augmentation methods included changing image attributes, randomly cropping, rotating, and flipping those images. The UNet structure was improved by designing eight different multi-scale image feature extraction modules. The module structure mainly included two aspects: Image convolution and feature fusion. The convolution improvement included convolutional block attention module (CBAM), depthwise separable convolution (DP Conv), and convolution (Conv). In terms of feature fusion methods, improvements could be divided into concatenation and addition. Subsequently, ablation tests were conducted based on a single dataset, data augmentation, and random loading of model weights, and the optimal multi-scale feature extraction module was selected and compared with the original UNet. Similarly, a single dataset, data augmentation, and random loading of model weights were used to compare and validate the advantages of the improved model with the PSPNet, SegNet, and DeeplabV3Plus algorithms. The improved model used pre-trained weights from a single dataset to load and train the model based on mixed datasets and data augmentation, further improving the model's generalization ability and root segmentation ability. [Results and Discussions] The results of the ablation tests indicated that Conv_ 2+Add was the best improved algorithm. Compared to the original UNet, the mIoU, mRecall, and root F1 values of the model increased by 0.37%, 0.99%, and 0.56%, respectively. And, comparative experiments indicate Unet+Conv_2+Add model was superior to the PSPNet, SegNet, and DeeplabV3Plus models, with the best evaluation results. And the values of mIoU, mRecall, and the harmonic average of root F1 were 81.62%, 86.90%, and 77.97%, respectively. The actual segmented images obtained by the improved model were more finely processed at the root boundary compared to other models. However, for roots with deep color and low contrast with soil particles, the improved model could only achieve root recognition and the recognition was sparse, sacrificing a certain amount of information extraction ability. This study used the root phenotype evaluation software Rhizovision to analyze the root images of the Unet+Conv_2+Add improved model, PSPNet, SegNet, and DeeplabV3Plu, respectively, to obtain the values of the four root phenotypes (total root length, average diameter, surface area, and capacity), and the results showed that the average diameter and surface area indicator values of the improved model, Unet+Conv_2+Add had the smallest differences from the manually labeled indicator values and the SegNet indicator values for the two indicators. Total root length and volume were the closest to those of the manual labeling. The results of transfer learning experiments proved that compared with ordinary training, the transfer training of the improved model UNet+Conv_2+Add increased the IoU value of the root system by 1.25%. The Recall value of the root system was increased by 1.79%, and the harmonic average value of F1 was increased by 0.92%. Moreover, the overall convergence speed of the model was fast. Compared with regular training, the transfer training of the original UNet improved the root IoU by 0.29%, the root Recall by 0.83%, and the root F1 value by 0.21%, which indirectly confirmed the effectiveness of transfer learning. [Conclusions] The multi-scale feature extraction strategy proposed in this study can accurately and efficiently segment roots, and further improve the model's generalization ability using transfer learning methods, providing an important research foundation for crop root phenotype research.