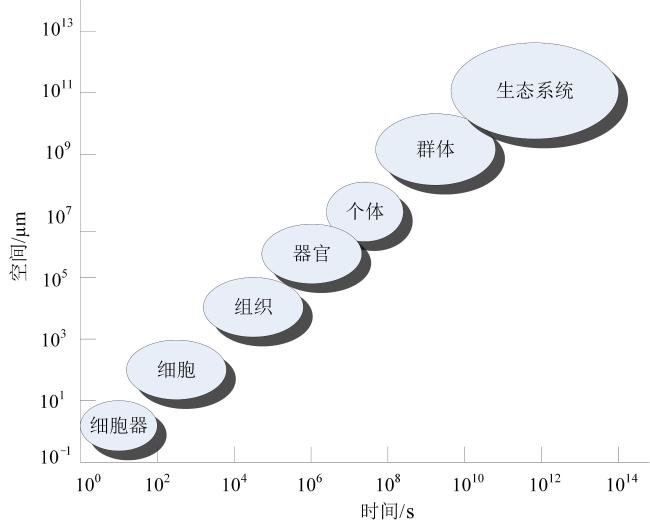
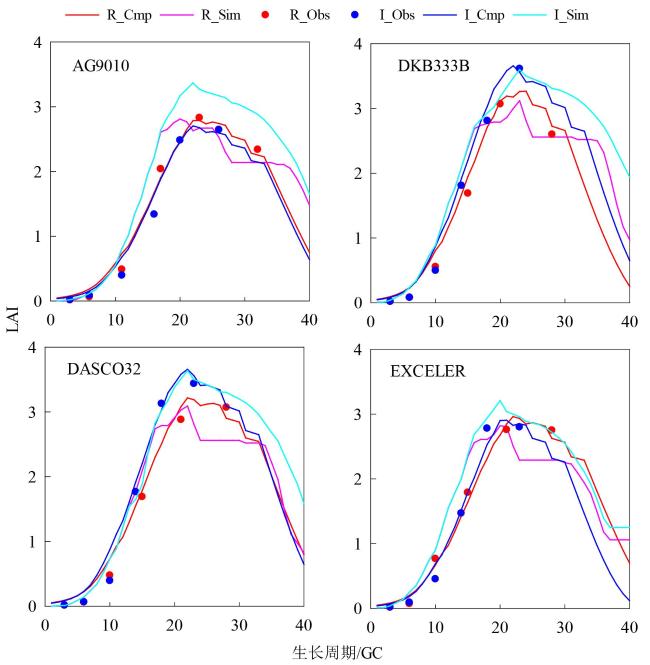
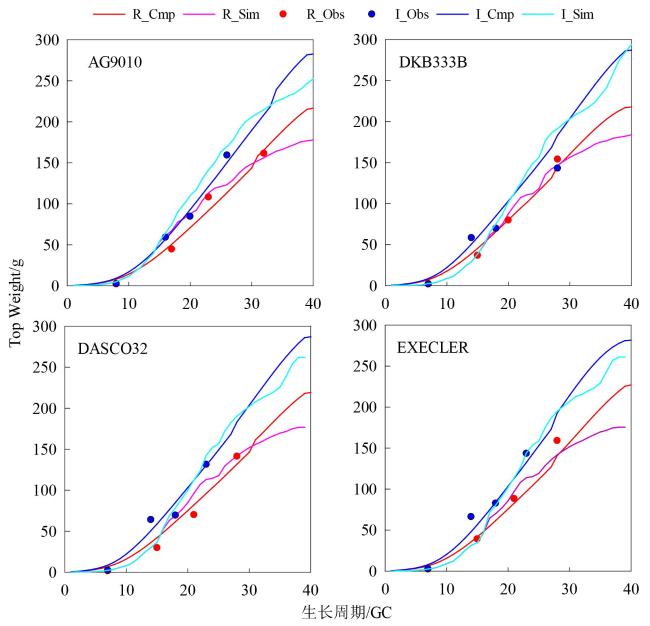
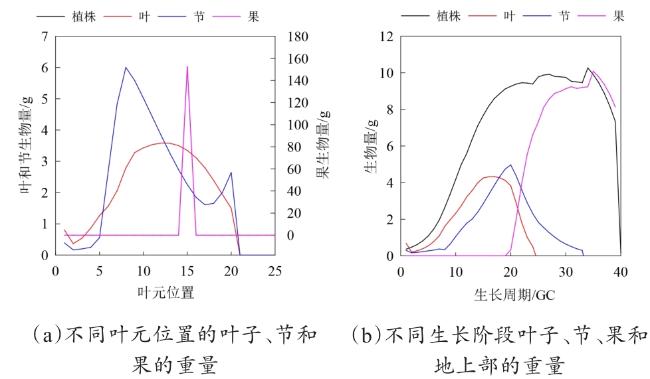
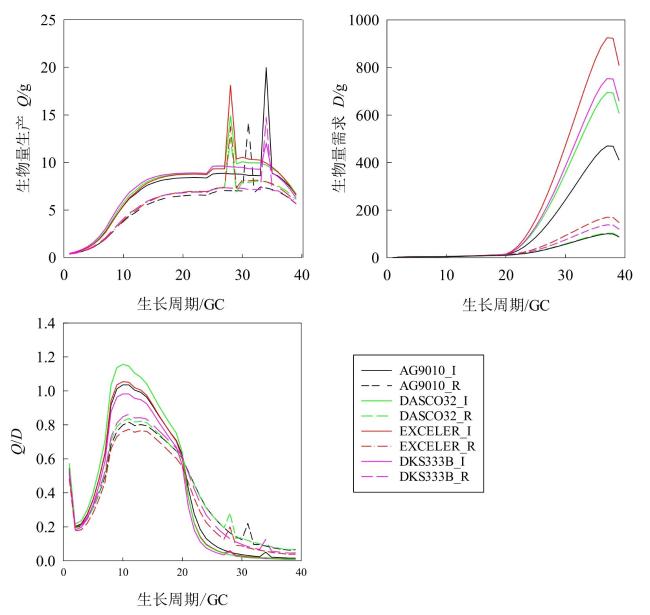
作物模型可分为描述性模型(Descriptive Model)和解释性模型(Explanatory Model)。前者又称为统计模型、回归模型、经验模型或黑箱模型,直接建立输入与输出之间的关系,不关心内在形成机理。后者又称为基于过程的模型(Process-Based Model),是指能定量地和动态地描述作物生长、发育和产量形成过程及其对环境反应的计算机模拟程序
[6],通常是在群体水平对作物生长的描述。常见的基于过程的作物模型,如CERES
[7]、TomSim
[8]、STICS(Simulateur mulTIdisciplinaire pour les Cultures Standard)
[9]和APSIM(Agricultural Production Systems sIMulator)
[10]等,模拟作物的光合作用过程、同化物的分配过程等,主要通过叶面积指数(Leaf Area Index,LAI)来预测每平方米的生物量产量,同时考虑辐射、温度和基本投入(灌溉和化肥等)的影响。这类模型大多将不同类型的器官作为一个整体考虑,关注作物的总果实重量、叶重等,难以描述作物个体内结构的变化及其对产量的影响。以作物模型为核心的农业技术转移支持决策系统(Decision Support System for Agrotechnology Transfer,DSSAT)以天、日为步长模拟作物生长,按照作物生育期详细描述作物生长发育过程,包括发芽到开花、叶片出现、开花时期、籽粒灌浆、生理成熟和收获等,并且可响应许多因素,包括作物遗传特性、管理措施、环境、氮素和水分的胁迫、病虫害等,主要用于农业试验分析、农业产量预报、农业生产风险评估、气候对农业的影响评价等
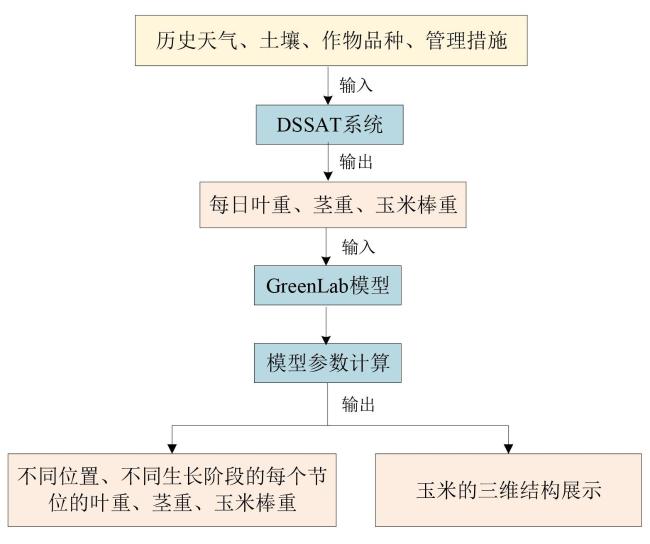
[2]。