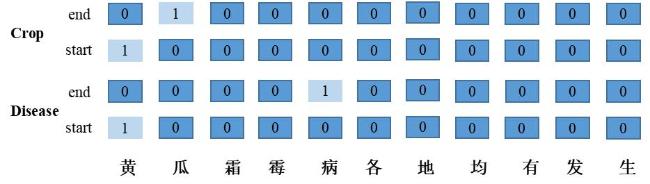
[Objective] With the development of agricultural informatization, a large amount of information about agricultural diseases exists in the form of text. However, due to problems such as nested entities and confusion of entity types, traditional named entities recognition (NER) methods often face challenges of low accuracy when processing agricultural disease text. To address this issue, this study proposes a new agricultural disease NER method called RoFormer-PointerNet, which combines the RoFormer pre-trained model with the PointerNet baseline model. The aim of this method is to improve the accuracy of entity recognition in agricultural disease text, providing more accurate data support for intelligent analysis, early warning, and prevention of agricultural diseases. [Methods] This method first utilized the RoFormer pre-trained model to perform deep vectorization processing on the input agricultural disease text. This step was a crucial foundation for the subsequent entity extraction task. As an advanced natural language processing model, the RoFormer pre-trained model's unique rotational position embedding approach endowed it with powerful capabilities in capturing textual positional information. In agricultural disease text, due to the diversity of terminology and the existence of polysemy, traditional entity recognition methods often faced challenges in confusing entity types. However, through its unique positional embedding mechanism, the RoFormer model was able to incorporate more positional information into the vector representation, effectively enriching the feature information of words. This characteristic enabled the model to more accurately distinguish between different entity types in subsequent entity extraction tasks, reducing the possibility of type confusion. After completing the vectorization representation of the text, this study further emploied a pointer network for entity extraction. The pointer network was an advanced sequence labeling approach that utilizes head and tail pointers to annotate entities within sentences. This labeling method was more flexible compared to traditional sequence labeling methods as it was not restricted by fixed entity structures, enabling the accurate extraction of all types of entities within sentences, including complex entities with nested relationships. In agricultural disease text, entity extraction often faced the challenge of nesting, such as when multiple different entity types are nested within a single disease symptom description. By introducing the pointer network, this study effectively addressed this issue of entity nesting, improving the accuracy and completeness of entity extraction. [Results and Discussions] To validate the performance of the RoFormer-PointerNet method, this study constructed an agricultural disease dataset, which comprised 2 867 annotated corpora and a total of 10 282 entities, including eight entity types such as disease names, crop names, disease characteristics, pathogens, infected areas, disease factors, prevention and control methods, and disease stages. In comparative experiments with other pre-trained models such as Word2Vec, BERT, and RoBERTa, RoFormer-PointerNet demonstrated superiority in model precision, recall, and F1-Score, achieving 87.49%, 85.76% and 86.62%, respectively. This result demonstrated the effectiveness of the RoFormer pre-trained model. Additionally, to verify the advantage of RoFormer-PointerNet in mitigating the issue of nested entities, this study compared it with the widely used bidirectional long short-term memory neural network (BiLSTM) and conditional random field (CRF) models combined with the RoFormer pre-trained model as decoding methods. RoFormer-PointerNet outperformed the RoFormer-BiLSTM, RoFormer-CRF, and RoFormer-BiLSTM-CRF models by 4.8%, 5.67% and 3.87%, respectively. The experimental results indicated that RoFormer-PointerNet significantly outperforms other models in entity recognition performance, confirming the effectiveness of the pointer network model in addressing nested entity issues. To validate the superiority of the RoFormer-PointerNet method in agricultural disease NER, a comparative experiment was conducted with eight mainstream NER models such as BiLSTM-CRF, BERT-BiLSTM-CRF, and W2NER. The experimental results showed that the RoFormer-PointerNet method achieved precision, recall, and F1-Score of 87.49%, 85.76% and 86.62%, respectively in the agricultural disease dataset, reaching the optimal level among similar methods. This result further verified the superior performance of the RoFormer-PointerNet method in agricultural disease NER tasks. [Conclusions] The agricultural disease NER method RoFormer-PointerNet, proposed in this study and based on the RoFormer pre-trained model, demonstrates significant advantages in addressing issues such as nested entities and type confusion during the entity extraction process. This method effectively identifies entities in Chinese agricultural disease texts, enhancing the accuracy of entity recognition and providing robust data support for intelligent analysis, early warning, and prevention of agricultural diseases. This research outcome holds significant importance for promoting the development of agricultural informatization and intelligence.