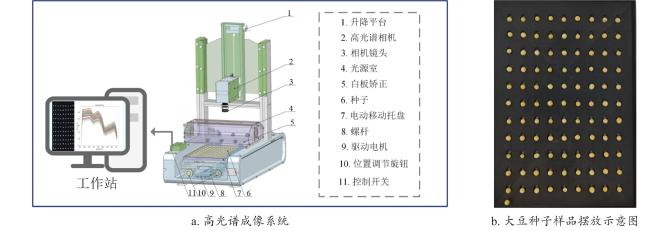
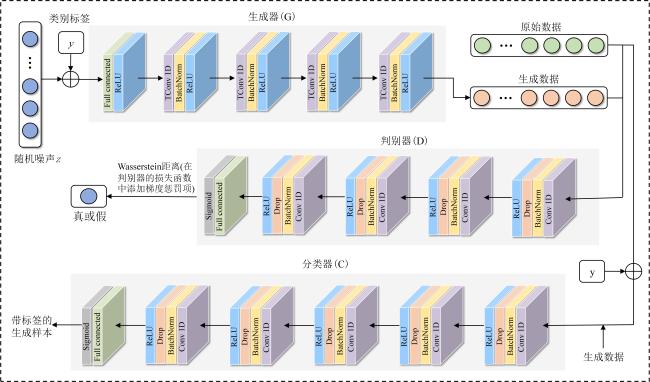
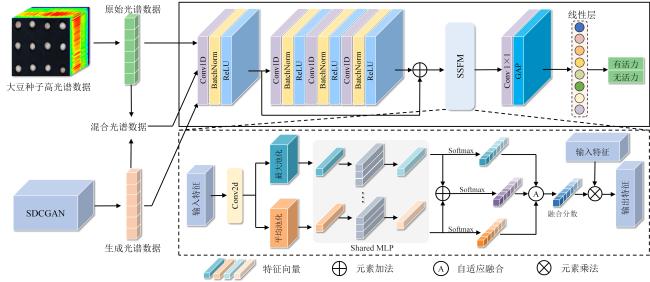
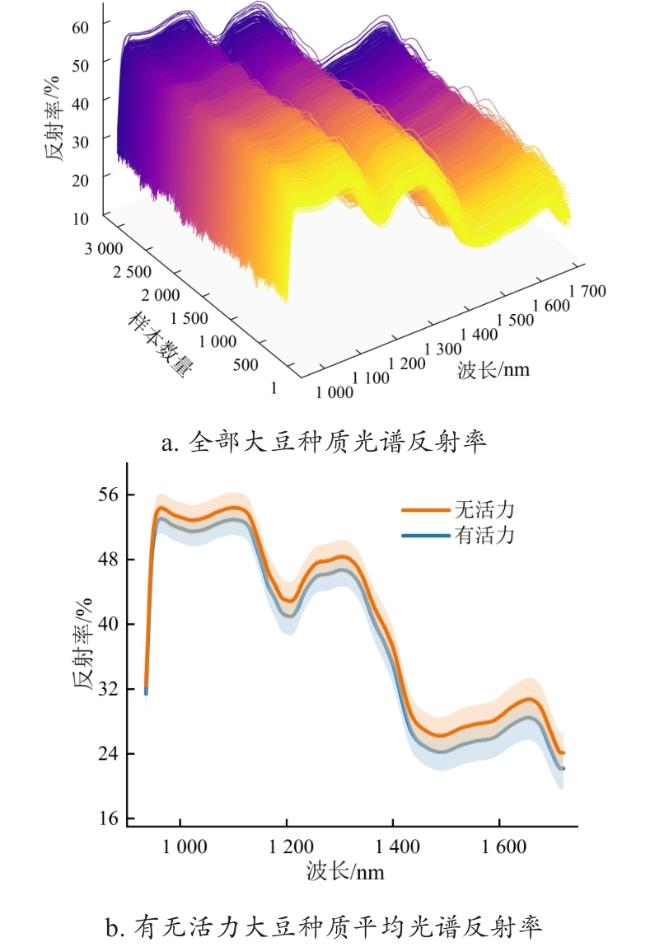
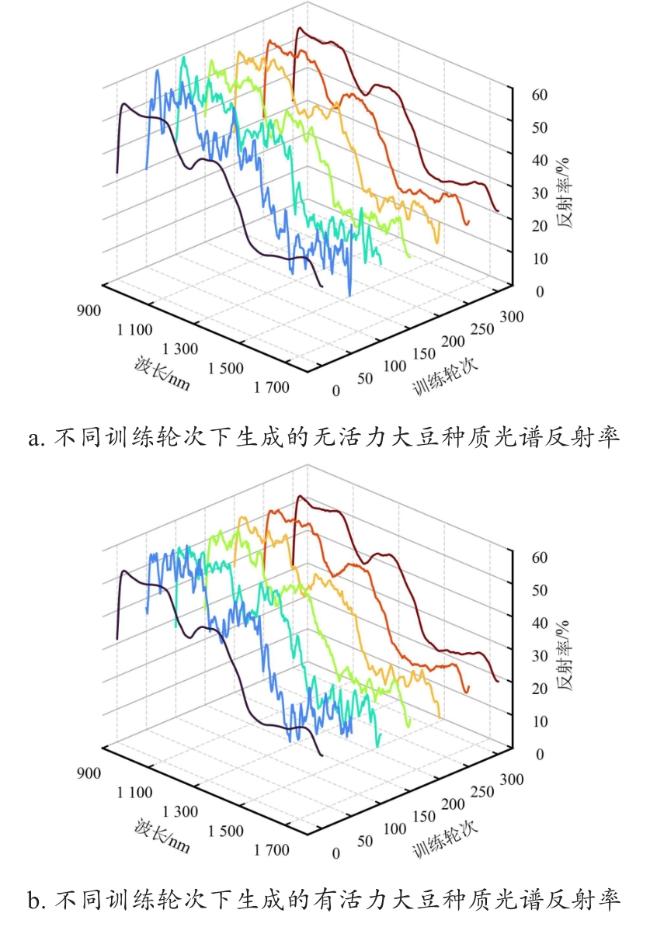
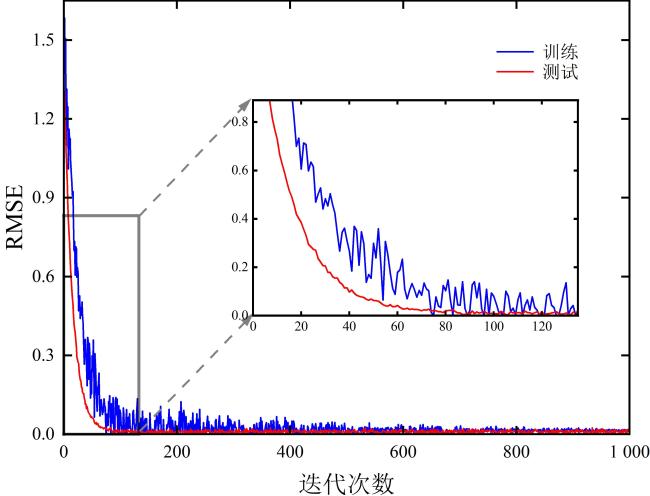
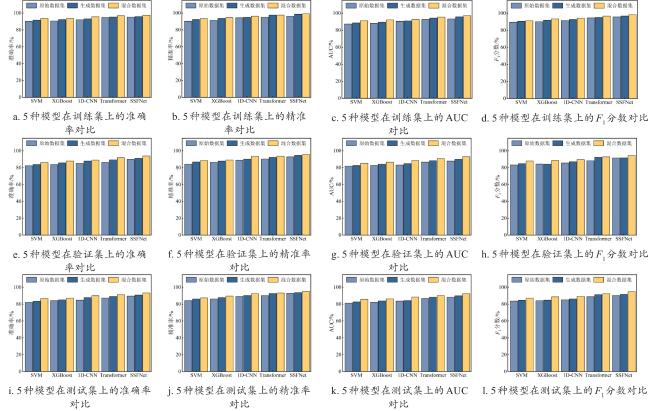
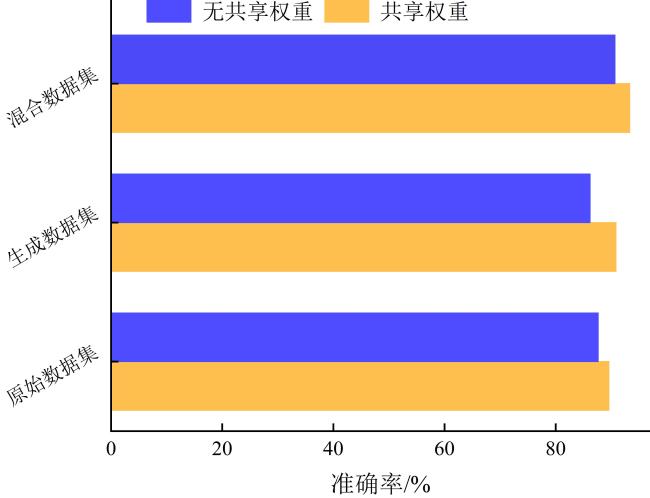
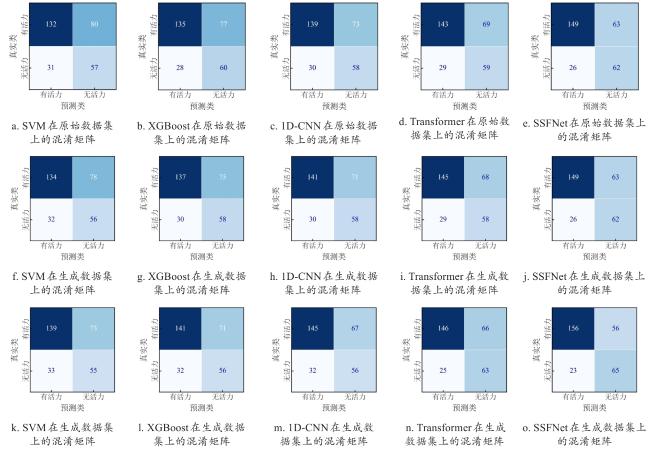
[Objective] Germplasm resources are regarded as the "chips" of high-quality breeding, and evaluating the viability of soybean germplasm is essential for ensuring the secure preservation of genetic resources and promoting the healthy development of the soybean industry. Traditional viability detection methods are time-consuming, labor-intensive, and seed-consuming, highlighting the urgent need for non-destructive, intelligent, and high-throughput detection technologies. Hyperspectral imaging combined with deep learning offers a promising approach for the rapid, non-destructive assessment of soybean germplasm viability. Compared to artificially aged samples, naturally aged samples more accurately reflect the substance changes associated with the decline in germplasm viability. However, the imbalance in the number of viable and non-viable samples limits the generalization performance of viability prediction models. [Methods] In order to address the aforementioned challenges, this study proposed a semi-supervised deep convolutional generative adversarial network (SDCGAN) to generate high-quality hyperspectral data with associated viability labels. The SDCGAN framework consisted of three main components: a generator, a discriminator, and a classifier. The generator progressively transformed low-dimensional latent representations into hyperspectral data. This was achieved through four one-dimensional transposed convolutional layers, ensuring the output matched the dimensionality of real spectra. The discriminator adopted an optimization strategy based on the wasserstein distance, replacing the Jensen-Shannon divergence used in traditional GANs, thereby mitigating training instability and gradient vanishing. Additionally, a gradient penalty term was introduced to further stabilize model training. In the classifier, a unilateral margin loss function was employed to penalize only those samples near the decision boundary, effectively avoiding overfitting on well-separated samples and improving training efficiency. Furthermore, this study developed a spectral score fusion network (SSFNet) to enable hyperspectral-based detection of soybean seed viability. SSFNet comprised two core modules: a spectral residual network and a spectral score fusion module. The spectral residual network extracted shallow-level features from the hyperspectral data, capturing local patterns within spectral sequences. The spectral score fusion module adaptively reweighted spectral channels to emphasize viability-related features and suppress redundant noise. Finally, the performance of the SDCGAN-generated spectra was evaluated using root mean square error (RMSE), while the viability detection performance of SSFNet was assessed using test accuracy, precision, area under the curve (AUC), and F1-Score. [Results and Discussions] In the performance analysis of SDCGAN, the model progressively learned and captured the key spectral features that distinguished viable and non-viable soybean seeds during the training process. The generated spectra gradually evolved from initial noisy fluctuations to smoother curves that closely resembled real spectra, demonstrating strong nonlinear modeling capability. Compared to other generative adversarial models, SDCGAN achieved the best performance in enhancing viability detection, and its generated data exhibited low error characteristics in RMSE analysis. By applying SDCGAN for data augmentation, three types of datasets were constructed: original spectra, generated spectra, and mixed spectral dataset. When using the MSC-SG-SS preprocessing strategy, SSFNet achieved the highest viability detection accuracies across all three datasets, reaching 89.50%, 90.83%, and 93.33%, respectively. In comparison with other viability detection models, SSFNet consistently outperformed alternative algorithms in all four evaluation metrics across all datasets. Particularly on the mixed dataset, SSFNet demonstrated the best performance, achieving a test accuracy of 93.33%, precision of 95.17%, AUC of 92.58%, and F1-Score of 94.83%. Notably, all models trained on the mixed dataset containing SDCGAN-generated samples achieved better performance than those trained on either original or generated datasets alone. This improvement was likely due to the increased sample diversity and balanced class distribution in the mixed dataset, which provided more comprehensive viability-related features, facilitated model convergence, and reduced overfitting. In transfer experiments, SSFNet also exhibited superior generalization capability compared to four baseline algorithms: SVM, XGBoost, 1D-CNN, and Transformer, achieving the highest classification accuracy of 73.67% on the mixed dataset. [Conclusions] This study constructs an integrated SDCGAN-SSFNet framework for robust viability detection of naturally aged soybean germplasm under imbalanced sample conditions. The SDCGAN component accurately learns the underlying distributional characteristics of real hyperspectral data from soybean seeds and generates realistic synthetic samples, effectively augmenting the spectral data of non-viable seeds and improving data diversity. Meanwhile, SSFNet explores inter-band spectral correlations to adaptively enhance features that are highly relevant to viability classification while effectively suppressing redundant and noisy information. This integrated approach enables rapid, nondestructive, and high-precision detection of soybean seed viability under challenging sample imbalance scenarios, providing an efficient and reliable method for seed quality assessment and agricultural decision-making.